我们研究基因组时,在生物学上的研究对象是sequence,即ATGC序列。与sequence不同,genomic feature(在Bioconductor中对应GRange/IRange对象)不显示ATGC内容,而着重强调各种feature (如gene, transcript, UTR, promoter, exon, intron, SNV, INDEL等功能元件)在(特定版本)基因组上的位置,包括染色体、正负链、起止位置等。因此,feature是sequence的数字化表示;一个特定的feature与一段sequence是一一对应的。这样可以把一个自然现象加以数学化,便于统计分析。
操作和转换Sequence和Feature有很多方式,但个人喜欢命令行方式,这要比GUI程序更有自由感。
1.Sequence (以fasta文件形式存在)
获得:
- 任意fasta文件
从基因组提取:
- samtools faidx hg38 chr1:10000-20000
- getdna: 自己写的bash脚本,samtools仅仅是通过chr:start-end方式获得,但getdna可以通过多种方式提取序列。而且根据chr分别加载不同染色体的基因组序列,可以节省内存。123getdna chr start widthgetdna w chr start endgetdna f chr middle flank
随机序列emboss中makenucseq和makeprotseq
定位: 本质是序列比对
blast和blat
emboss的needle: Needleman-Wunsch global alignment of two sequences
emboss的water: Smith-Waterman local alignment of sequences
emboss的dreg,preg,fuzznuc,fuzzprot
显示:
cat x.fasta
dispseq x.fasta: 自己写的bash脚本,方便计算位置
2.Feature/Range (常以bed/gtf/gff3等文件形式存在)
获得:
- 基因组注释库,如bed、gtf、gff3等格式
- vcf文件,可以转化为bed等格式
- 手写bed
定位: 本质是overlap/基因组注释流程
bedtools
12345678910bedtools基本用法: bedtools -a -b; map a to b, so report annotation from abedtools intersect -a A.bed -b B.bed # a中与b相交的feature,但只显示相交部分的坐标bedtools intersect -a A.bed -b B.bed -wa # a中与b相交的feature,显示a的坐标bedtools intersect -a A.bed -b B.bed -wb # a中与b相交的feature,显示b的坐标bedtools intersect -a A.bed -b B.bed -wa -wbbedtools intersect -a A.bed -b B.bed -c # a中与b相交的个数bedtools intersect -a A.bed -b B.bed -v # a中没有与b相交的featurebedtools intersect -a A.bed -b B.bed -f 0.50 -r -wa -wbbedtools window -a A.bed -b B.bed -w 200 #把A上下游各加上200bp后,再intersect Bbedtools window -a A.bed -b B.bed -l 200 -r 20000annovar | oncotator等注释软件
显示:
- IGV等可视化软件
- igvseq: 自己写的脚本,调用IGV,以多种方式输入feature123456igvseq chrigvseq chr begin widthigvseq w chr begin end # w means widthigvseq f chr middle flank # f means flankingigvseq r chr begin end # r means regionigvseq l file # l means loading file
3.转换
Sequence和Feature(Range)转换参考以上描述。
PS: 0-based和1-based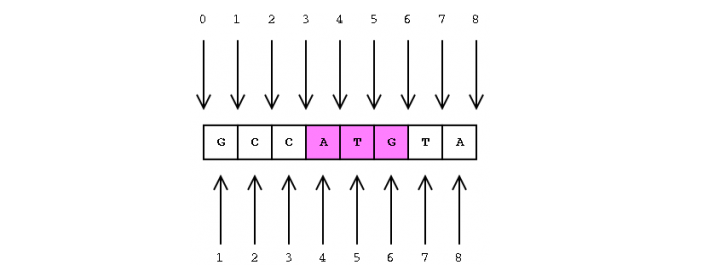
0-based and half-open file format: [start, end)
bed, bedgraph, wig, bam, dbSNP
1-based and fully-closed file format: [start, end]
bigwig, gff, vcf, sam
UCSC的Tables使用的是0-based;
UCSC的Genome Browser使用的是1-based;


