本文总结了PyMC3里可用的分布,包括连续型分布和离散型分布。给出了它们的概率密度公式、统计指标和分布图,以方便选择合适的先验分布。资料来源于https://docs.pymc.io/api/distributions.html。

Yang's Garden
记录我的所见、所闻、所学、所得 Email:sunnysean@qq.com
概率编程(Probabilistic programming)的PyMC3实现
概率编程中的概率是指概率推理,特指贝叶斯方法里的概率及其求解;编程是指通过计算机编程语言(如Python)去实现建模和求解。其中关键是变分推断(Variational inference)技术。PyMC3是Python中实现概率编程的模块,它利用了新一代的MCMC抽样算法(如NUTS),因而计算速度快,使得概率编程容易实现。本文举了多个例子,其中线性回归的例子中详细介绍了pymc3的用法。
基因突变的寻找和功能评估
二代测序使寻找基因突变变得更容易了,科研者可以很方便地获得任何个体的整个基因组范围内的突变。但是医学问题并没有因此得到解决,因为存在大量无关紧要的突变,这是基因多样性的一种表现,而非病理性的异常。此外对于肿瘤疾病而言,基因组上存在大量的突变,筛选关键突变成为重中之重,无论对于病因学还是基因靶向治疗而言都是必需的。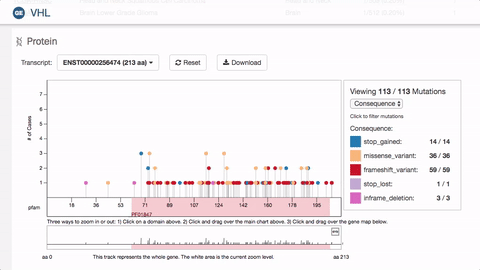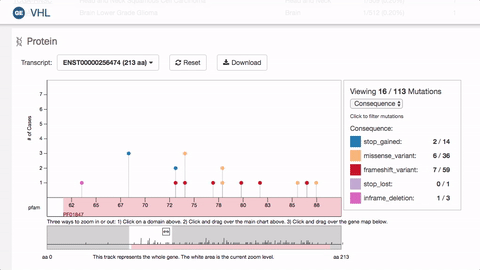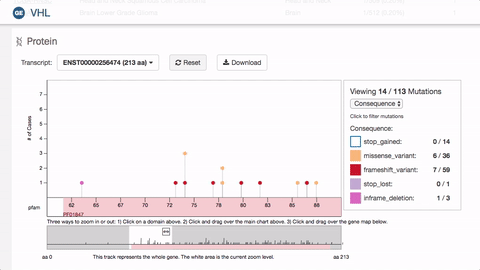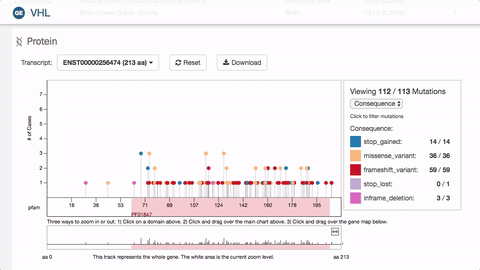
GATK4简明用法
GATK4是最新的GATK版本,它在算法上进行了优化,运行速率得到提高,而且整合了picard。GATK4依然是用java 语言开发的,但使用方式上更加人性化,比如所有命令都是gatk cmd方式,这里的cmd是任何可以用的cmd。GATK4 的最佳实践给出了5套pipeline: Germline SNP/Indel, Somatic SNV/Indel, RNAseq SNP/Indel, Germline CNV, Somatic CNV。本文是前段时间参与Broad和Intel中国在北京的培训班上的精简记录,供自己参考用,主要是我所关注的SNV/Indel。
深度学习在基因组学中的应用
深度学习方法,如CNN、RNN、Auto-Encoder等,已经广泛用于对图像、语音、文本等数据进行机器学习。但是涉及DNA序列的基因组数据显然与上述数据类型不同。此外,深度学习是“黑箱”模式;相反,基因组序列有明显的生物学意义。本文总结了当前基因组学研究中如何针对DNA序列数据进行深度学习,以管中窥豹。
蛋白质动力学:一门与分子生物学互补的知识
蛋白质是生物体结构和功能的基本单位。研究蛋白质,有两大类不同研究的思路。一种是分子生物学研究,其研究对象为大量的蛋白质分子(例如western blotting中μg级别的上样量,ELISA的ng/ml级别检出限<大概也是μg级别>)。它观察蛋白质分子群体的平均行为,如研究信号通路中上游蛋白引起下游蛋白的激活或失活等。另一类是分子动力学研究,其研究对象为蛋白质分子个体,既有计算机模拟,也有实验方法(如核磁共振动力学),后者是前者的补充。
量子生物学与《量子生物学入门》
量子生物学试图用量子理论(量子化学)解释一些生命现象。但现在许多”民科”和非科学的东西都打着量子的旗号。即便是科学的东西,如已经出版的那些量子生物学的科普书,多数是脑洞大开的想象。毕竟生物学的基本功能单位都是分子水平的,而以原子和电子为研究对象的量子理论/量子化学远不能解释复杂的、自我演化的生命现象。不过,在探索生物大分子功能背后的微观机制时,量子化学对一些特定的生物机制能提供一些解释。后者才是真正有科学意义的量子生物学。此时的量子生物学,属于计算化学的内容,本质是用化学规律解释生命规律。这与一些脑洞大开的、玄学性质的”量子”没有联系。
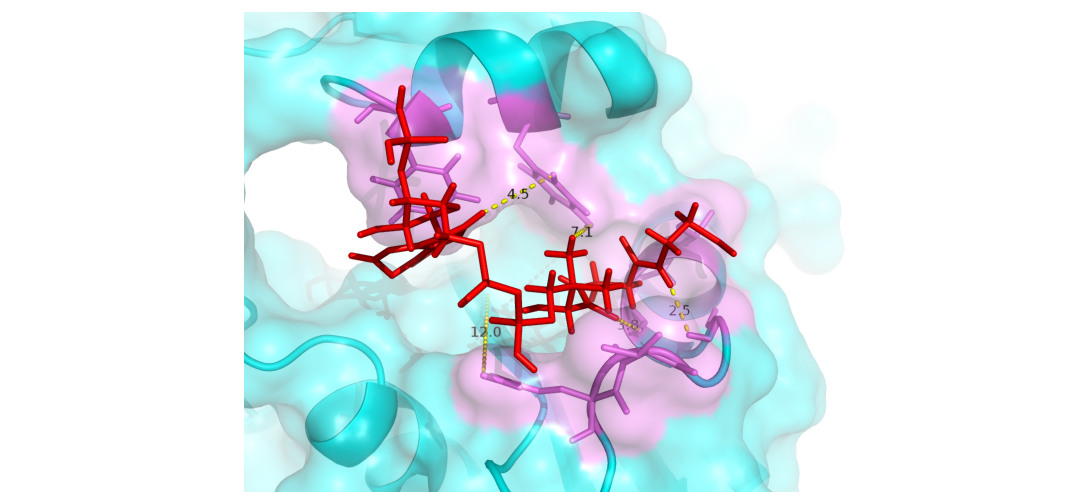
蛋白-配体结合:联合使用分子对接和分子动力学模拟
研究蛋白质和配体的结合模式,最重要的两个计算生物学方法是分子对接(Molecular Docking)和分子动力学模拟(Molecular Dynamic Simulation, MD)。两者各有特点,相互补充。